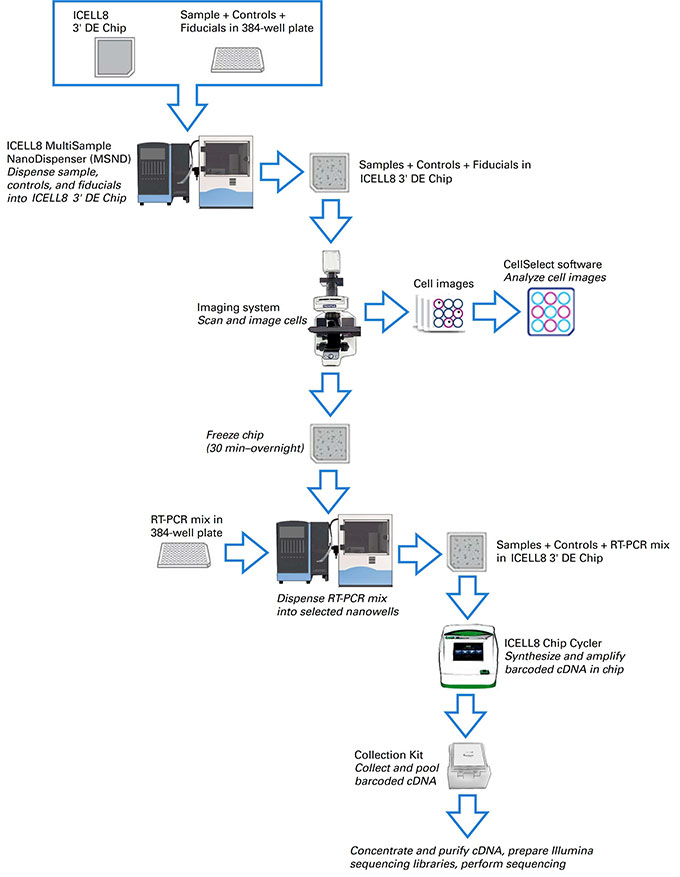
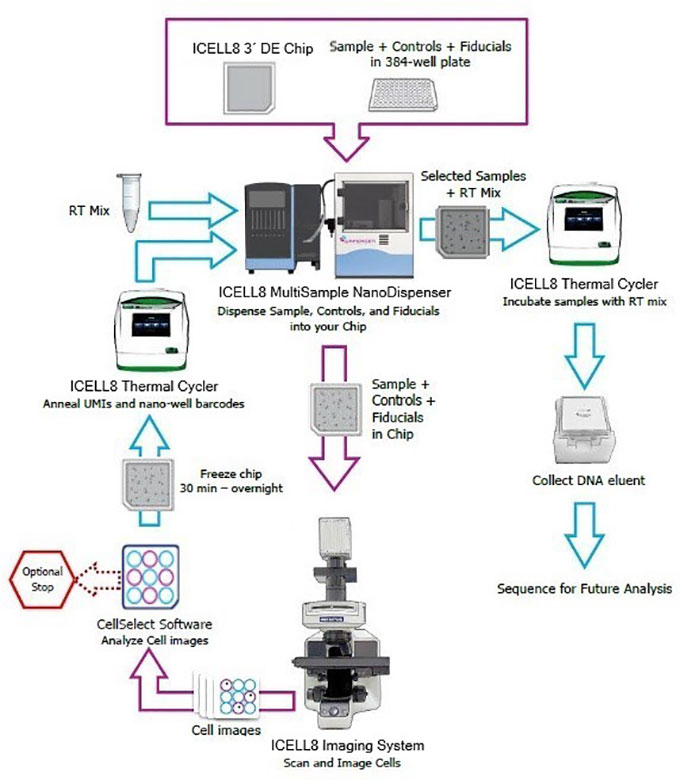
| 自动化高通量的单细胞差异表达分析 | ||||||
| 品牌 | Code No. | 产品名称 | 包装量 | 价格(元) | 说明书 | 数量 |
| Clontech | 640164 | ICELL8® Chip and Reagent 3’DE Kit | 1 Rxn | 询价 | |
  |
| Clontech | 640165 | ICELL8® Chip and Reagent 3’DE Kit | 3 Rxns | 询价 | |
  |
| Clontech | 640166 | ICELL8® Chip and Reagent 3’DE Kit | 5 Rxns | 询价 | |
  |
| Clontech | 640167 | ICELL8® 3’DE Reagent Kit | 1 Rxn | 询价 | |
  |
| Clontech | 640143 | ICELL8® 3’DE Chip | 1 Chip | 询价 | |
  |
| Clontech | 640048 | ICELL8® Collection Kit | 1 Pkg | 询价 | |
  |
| Clontech | 640109 | ICELL8® Loading Kit | 1 Pkg | 询价 | |
  |
| Clontech | 640005 | ICELL8® 3’DE for UMI Reagent Kit | 1 Rxn | 询价 | |
  |
| Clontech | 640192 | ICELL8® 384-Well Source Plate and Seal (5/pack) | Each | 询价 | |
  |
收藏产品 加入购物车
| 高通量、高品质、更经济的单细胞3’端转录组分析解决方案 | ||||||||||||||||||||||||||||||
|